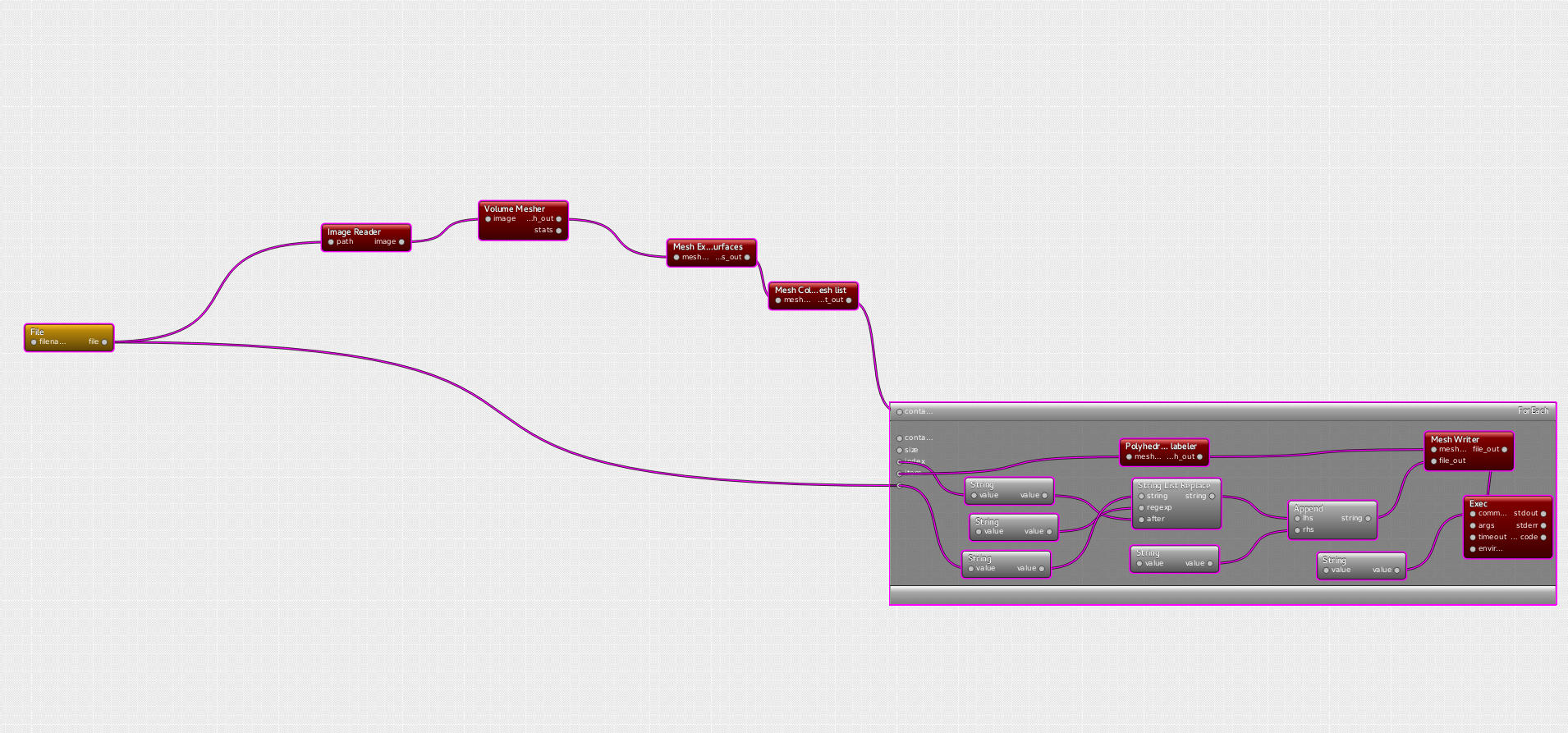
Visual programming framework of dtk enables to create very complex pipelines in a very short time.
The case at stake here is provided by provided by Shuman Jia, PhD at Asclepios and it involves image processing algorithms and discrete geometry.
<!–more–
##Heart Surface Extraction pipeline
The purpose of this pipeline is to generate a triangulated surface mesh representing the external surface of a heart, from a mask given as a “.mha” image. Then, from this surface mesh, one wants automatically carrying out a segmentation to isolate the main features of the heart: ventricule, superior vena cava/ ascending aorta, inferior vena cava/descending aorta, and the aortic arch.
The constraint for the mesher is a relatively small number of triangles (<30k) while keeping both a good representation of the heart and a good quality of triangles (here one wants preserve isotropy in R3).
Using interfaces provided by dtk, the pipeline therefore resorts to algorithms from the ITK library (https://itk.org/) for the medical imaging and algorithms from Cgal library (http://www.cgal.org/) for the geometry.
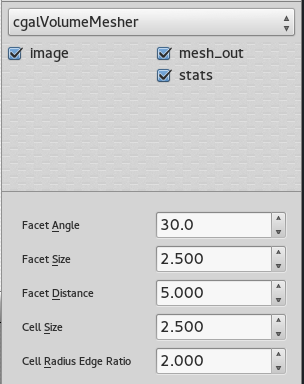
Volume mesher
The mesher used in this case is the Cgal volume mesher, which produces good quality elements. The size of these elements can be easily modified through the interface of dtk-visual-programming, as well as the different properties of the mesh.
 |
 |
:—: | :—: |
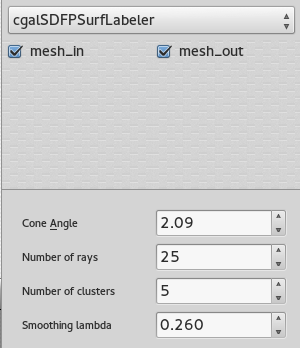
SUrface extraction and labelling
Once the volume is tessellated, the tool Mesh extract surfaces is invoked to recover only the external surface (possibly external surfaces). This surface goes through the Polyhedral labeler, in order to be segmented into an appropriate number of connex components, based on its volume properties. For more information on the different parameters available, consider reading : Consistent mesh skeletonisation.
The number of clusters fixed in the GUI is an approximation of the final number of parts.
Since the CGAL mesher works as a probe slides_L3 (which intersects the appropriate level set, f(x,y,z)=0 for a binary image), it is appropriate to produce multi resolution surfaces of the same heart. It will be possible using the CGAL library, to keep track of the distance between the high resolution model and the lower resolution model.
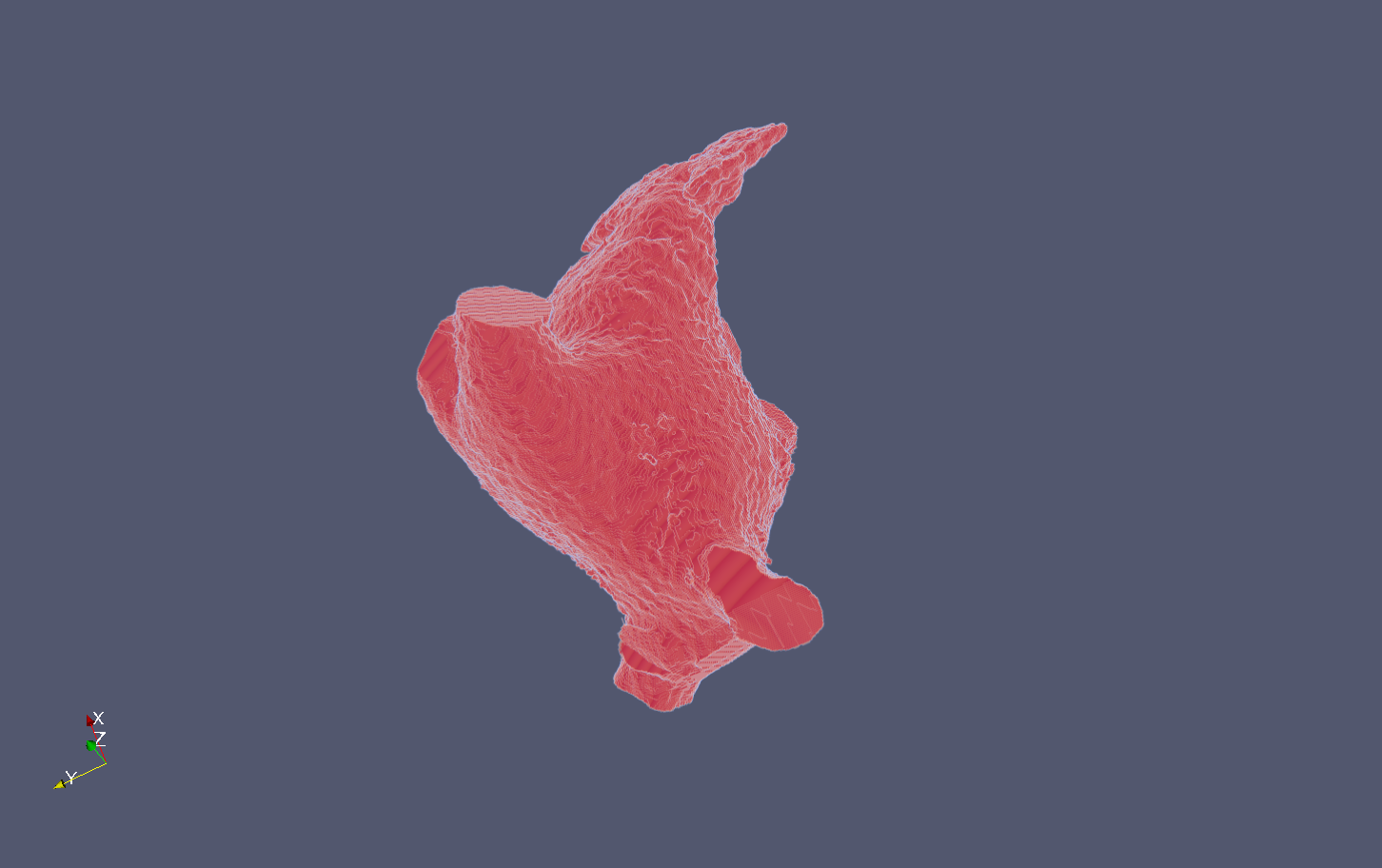
Results
The following snapshots display the initial image (.mha) and the result of the process (.vtu) :
La programmation visuelle de dtk permet de créer rapidement des pipelines qui enchainent des opérations non triviales.
Le pipeline qui nous intéresse ici est issu du travail de Shuman Jia, actuellement en thèse au sein de l’équipe Asclépios. Les opérations qui sont en jeux dans ce cas font appel à des algorithmes de traitement d’image et de géométrie discrète.
Pipeline d’extraction de surface cardiaque
L’objectif du pipeline est de générer un maillage surfacique triangulaire représentant la surface externe d’un coeur à partir d’un masque binaire fourni sous la forme d’une image “mha”. Puis il s’agit, à partir de ce maillage de surface, de réaliser une segmentation pour isoler les différentes parties du coeur : ventricule, la veine cave supérieure / aorte ascendante, la veine cave inférieure / aorte descendante, et l’arc aortique.
La contrainte pour le mailleur est de produire un nombre assez faible de triangles (moins de 30k) tout en gardant à la fois une bonne représentation du coeur et une bonne qualité de triangle (ici il faut préserver l’isotropie).
Le pipeline fait donc appel via les interfaces fournies par dtk à des algorithmes de la bibliothèque ITK (https://itk.org/) pour la partie traitement d’image et à des algorithmes de la bibliothèque Cgal (http://www.cgal.org/) pour la Géométrie.
Mailleur volumique
Le mailleur utilisé ici est donc celui de Cgal qui permet d’obtenir une bonne qualité de triangles. On peut facilement régler la taille de ces triangles ainsi que les différentes propriétés du maillage via l’interface de l’outil de programmation visuelle.
 |
 |
:—: | :—: |
Isolation des parties du coeur
À partir de cette triangulation volumique, on extrait la surface du coeur. Celle-ci est ensuite donnée à un outil de labélisation polyédrique (Polyhedral labeler) afin de segmenter (ou isoler) les parties connexes du coeur à partir de ces propriétés volumiques (cf. Consistent mesh skeletonisation pour plus d’information).
Le nombre de clusters demandé dans l’interface permet d’obtenir approximativement le nombre final de composantes connexes.
Le mailleur de Cgal fonctionne suivant un mécanisme de sondage en lançant des rayons qui intersectent une level-set (typiquement f(x,y,z)=0 pour une image binaire, cf. slides_L3). Cette propriété du mailleur permet de produire des surfaces multi-résolution d’un même coeur. Il est également possible d’utiliser Cgal pour garder une trace des distances entre les modèles hautes résolution et faible résolution.
Résultats
L’image de droite ci-dessous montre le maillage et les parties du coeur que l’on obtient à partir du masque binaire de gauche.